基本介绍
噬菌体展示技术(Phage Display)是一种强大的体外分子进化技术,通过将外源多肽或蛋白基因片段融合至噬菌体衣壳蛋白上,使其随噬菌体一起表达并暴露在外表面,从而构建大规模随机多样性的肽库或抗体库。该技术最早由 George P. Smith 于 1985 年提出,并在2018年,噬菌体展示研究的两位先驱,美国科学家乔治·史密斯(George P. Smith)和英国科学家格雷格·文特(Gregory P.Winter)荣获诺贝尔化学奖。
基本原理
噬菌体展示技术的原理在于通过基因工程手段,将目标蛋白或多肽的编码序列与噬菌体衣壳蛋白(如M13噬菌体的pIII或pVIII蛋白)基因融合,使得在噬菌体感染宿主细胞后,所产生的噬菌体颗粒不仅在表面展示出目标分子,同时内部还携带着相应的DNA信息,从而实现了展示分子与其编码基因的物理连接。在构建了一个包含大量不同分子变体的高多样性文库后,通过与特定靶标的亲和筛选,只有那些能以高亲和性和高特异性结合靶标的噬菌体能够被保留下来,经反复洗脱和扩增后,最终筛选出最优结合分子。这种技术为抗体发现、蛋白工程以及新型生物分子开发提供了一种高效、精准且便捷的体外分子筛选平台。
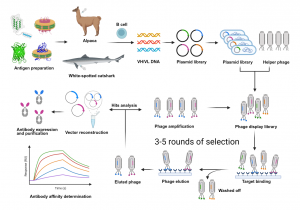
核心流程
1.文库构建:将外源 DNA 片段(如抗体可变区 VHH、随机肽库)插入 pIII 或 pVIII 基因,构建噬菌体展示载体(如 M13 phagemid)。
2.展示与感染:构建的载体转化大肠杆菌,与辅助噬菌体(Helper Phage)共感染,形成带有目标蛋白展示的 M13 噬菌体颗粒。
3.亲和筛选(Biopanning):将噬菌体库暴露于靶标(如抗原、受体等),洗去未结合的噬菌体,富集高亲和力结合子。
4.扩增与优化:回收结合的噬菌体,在大肠杆菌中扩增,并进行 3~5 轮筛选,提高亲和力与特异性。
5.下游应用:获得的高亲和力噬菌体可进一步进行抗体表达、蛋白工程优化或功能验证。
技术优势
1.多种蛋白表达系统,助力膜蛋白研究
搭建有完善的膜蛋白表达平台,特别针对 GPCR 等膜蛋白研发提供支持。团队拥有多年蛋白表达与纯化经验,能够高效、快速获取高质量抗原分子,确保筛选的可靠性。
2.智能分子模拟与结构分析
依托先进的分子模拟与结构分析平台,能够基于靶标抗原的序列和结构设计高复杂度文库。显著提升筛选成功率,助力获得高亲和力、高特异性的结合分子。
3.高效感受态细胞制备,超大文库构建
自主研发超级电转级感受态细胞,一次电转化即可实现 10⁹ 级别文库容量,满足大规模筛选需求。确保筛选的多样性和精准性,为优质分子的筛选奠定坚实基础。
4.高通量筛选平台,精准高效
结合 ELISA、SPR(表面等离子共振)等前沿筛选技术,实现快速筛选,显著提升实验效率。支持大规模、高灵敏度的筛选流程,助力精准分子筛选。
5.丰富的项目经验,应用广泛
具备丰富的筛选经验,可针对抗体、酶、受体-配体、蛋白互作等多种分子进行高效筛选。适用于生物医药、蛋白工程、药物研发等多个前沿领域。
6.超大规模人源抗体库,助力抗体药物开发
现有 10¹⁰ 级别人源天然抗体库,可直接用于人源化抗体筛选,大幅缩短抗体发现周期。为抗体药物开发提供高效、精准的筛选工具,加速从筛选到应用的转化进程。
案例
- GPRC5D 羊驼纳米抗体的筛选
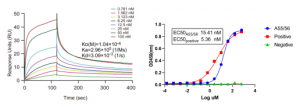
2.ROR1 羊驼纳米抗体的筛选

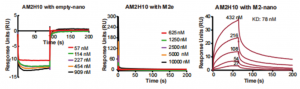
3.AM2鲨鱼纳米抗体的筛选
 合肥中科长木生物科技有限公司
合肥中科长木生物科技有限公司
您好!请登录